圖1 計算機仿真
蛋白質折疊過程需要大量計算
生物資訊學是結合基因蛋白質學及資訊科技的新興研究領域,其最終目標在發現新的生物認知、釐清細胞各階段的表現,以利人們對疾病和藥物使用有更精確的了解。故其現階段執行的任務有分析核酸序列、蛋白質序列、蛋白質結構、蛋白質家族及其生化反應等。
其中,需要資訊及半導體技術協助的研究包括:序列組合、序列分析、比較基因組學、計算基因遺傳學、基因認定、蛋白質三維結構分析、基因微數組晶片分析、分子演化、藥物設計等領域。
高性能計算設備方能應付大量運算需求
測序是基因研究的基礎,目前高通量基因測序設備及配套硬軟體均依賴進口,如何讓此核心技術生根關係著本土相關產業的發展。而此高性能的設備多用於以下兩個主要應用:
一、基因搜索及比對:將已知之 DNA 結構辨別標準輸入計算器內,可判斷樣本序列是否存在,以及此序列與不同物種間之 DNA 關聯性;
二、蛋白質摺疊仿真及計算器輔助藥物設計:利用重組或擴展已有的蛋白質是預防疾病和藥物設計的關鍵,因此計算器圖學技術與計算方法至關重要。以幾何、能量與活性等三大方向來探討如何使用最小能量原理來改進藥物設計中的分子對接過程之效能、加速藥物設計時程與降低研發成本是目前最迫切的課題。
為生物基因計算應用設計之 FPGA 系統
NVIDIA 的 CUDA 平台,其擁有通過 C 語言來控制 GPU 的開發環境,適合並行運算。在 CUDA 架構中,作 DNA 測序、短序列拼裝,可發揮並行計算之優勢,加速計算並生成三維基因圖譜。
Invitrogen 和 Active Motif 兩大生技公司也合作利用 FPGA 技術分析新一代的測序數據。 TimeLogic 生物計算系統由一至多片的 PCI Express x 1 之 FPGA 板卡組成,其整合FPGA 加速器和基因組學算法,能將新發現的數據和已知的基因進行快速比對,處理速度等同於普通 CPU 的 100 倍以上。
在要求更高的蛋白質摺疊仿真算法中,研究人員先以遺傳算法來加速接合位置的幾何搜尋;再以能量為重點,於實驗中使用李亞普諾夫函數中的穩定理論來降低接合位置數,以便進一步增進分子對接的效能;同時使用 NURBS 曲線中的插入頂點與權重調整來加速分子系統達到最小能量狀態;最後以各種不同的藥物受體模型來做計算機仿真計算,利用最小能量原理,判斷出接近全局能量最小區域的對接狀態之穩定度,並對其各種分子活性進行評估。
為了運用 FPGA 主晶片實現計算應用分子力場,以及配合 LYAPUNOV 指數求出降低分子對接的幾何位置數量與穩定度,我們提出以圖2中所示之系統架構來滿足更高層級研究人員所需。
如圖2所示,單片系統的主要功能是將高速輸入之數據,利用 4 組 GigE 網口,提供給後端 FPGA 進行序列比對處理。高硬件架構的核心部份為雙 DDR2 SODIMM 模塊之 Multi-Port Controller,內存的管理單元及總線設計是提升整體高速運算效能之關鍵。為了將序列比對時重要之 Linked List 數據結構以硬件 FPGA 實現,另外安排了特殊 ZBT (Zero Bus Turnaround) SRAM 以儲存 Pointer 之部份。使兩個讀寫總在線的子系統可以同時平行運作而不互相干擾,進而達到動態並行處理的效能。
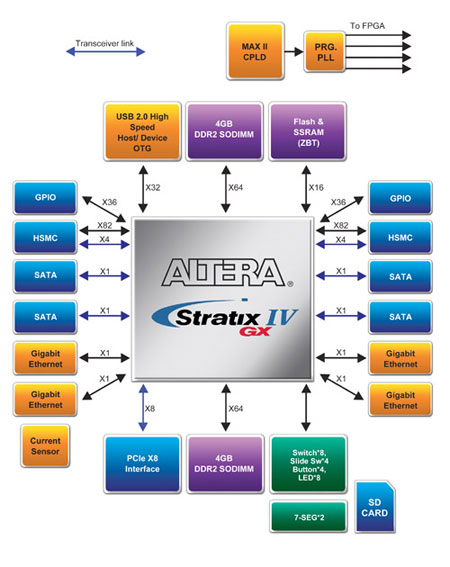
圖2 生物基因及蛋白質運算 FPGA 平台架構圖
由於 PC 主板以 DMA 方式收送軟體處數據,為了符合要求,系統以 PCI Express x 8 提供 16Gb/s 帶寬,系統並提供 2 個 HSMC 接頭做為板卡互連之高速接口,而 HSMC 接頭的關鍵頻寬由 8 對能承受 10Gb/s 之高速訊號線擔任。
以 HSPICE 仿真克服 10Gb/s 系統設計瓶頸
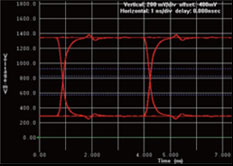
圖3 DDR2 / DDR3 之模擬結果
高速 FPGA 開發平台的訊號完整性是系統成功與否之關鍵。因此在設計過程中,我們運用包含 Hspice 在內的多種仿真分析工具,通過對具體問題進行分析來優化零配件選擇和設計折衷,如層迭結構、介電材料、訊號線拓樸結構、線長、線寬和阻抗匹配組件等,並根據仿真結果對設計進行調整,以便在設計時間內解決大多數的訊號完整性問題。
圖3為我們使用仿真分析阻抗匹配組件對 DDR2 地址訊號的影響,透過仿真可以看到終端匹配電阻的使用將使訊號擁有較少的 overshoot 與 undershoot。
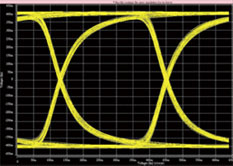
圖4 HSPICE 眼圖結果
圖5為最後依據多方研究人員需求,開發完成之 FPGA 平台。目前由台灣在蛋白質摺疊及藥物設計方面之專家進行算法加速上之測試。
對此平台細節有興趣之讀者,請參考 de4.terasic.com
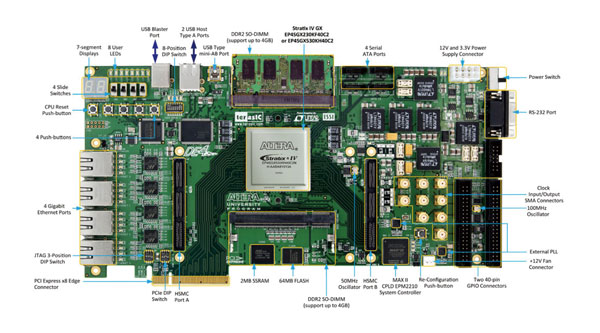
圖5 FPGA 系統實際成品照片
